コドン

mRNA分子に沿って一連のコドンを示している。各コドンは3ヌクレオチドからなり、一つのアミノ酸を指定している。
コドン(英: codon)とは、核酸の塩基配列が、タンパク質を構成するアミノ酸配列へと生体内で翻訳されるときの、各アミノ酸に対応する3つの塩基配列のことで、特に、mRNAの塩基配列を指す。DNAの配列において、ヌクレオチド3個の塩基の組み合わせであるトリプレットが、1個のアミノ酸を指定する対応関係が存在する。この関係は、遺伝暗号、遺伝コード(genetic code)等と呼ばれる。
ほぼ全ての遺伝子は厳密に同じコードを用いるから(#RNAコドン表を参照)、このコードは、しばしば基準遺伝コード(canonical genetic code)とか、標準遺伝コード(standard genetic code)、あるいは単に遺伝コードと呼ばれる。ただし、実際は変形コードは多い。つまり、基準遺伝コードは普遍的なものではない。例えば、ヒトではミトコンドリア内のタンパク質合成は基準遺伝コードの変形したものを用いている。
遺伝情報の全てが遺伝コードとして保存されているわけではないということを知ることは重要である。全ての生物のDNAは調節性塩基配列、遺伝子間断片、染色体の構造領域を含んでおり、これらは表現型の発現に寄与するが、異なった規則のセットを用いて作用する。これらの規則は、すでに十分に解明された遺伝コードの根底にあるコドン対アミノ酸パラダイムのように明解なものかも知れないし、それほど明解なものではないかも知れない。
目次
1 簡易解説・コドン
1.1 コドンはmRNA上にある
1.2 遺伝コードにおける塩基とアミノ酸の対応
1.3 生物種による利用コドンの偏り
2 遺伝コードの解読
3 遺伝コードを介して情報を伝達する
4 RNAコドン表
5 重要な特徴
5.1 塩基配列の読み枠
5.2 開始コドン、終止コドン
5.3 遺伝コードの縮重
6 標準遺伝コードの変形
7 遺伝コードの起源についての理論
8 引用文献
9 参考文献
10 関連項目
11 外部リンク
簡易解説・コドン
コドンはmRNA上にある
コドンは、厳密には実際のタンパク質の設計図として機能するmRNA中に存在している、アミノ酸1個に対応したヌクレオチドの塩基3個の配列のことを指す。RNAのヌクレオチドの塩基は、A(アデニン)、C(シトシン)、G(グアニン)、U(ウラシル)の4種類がある。そして、mRNA中の塩基の配列は、細胞で遺伝情報を保持しているDNAから転写されて作製されるので、コドンをDNA中の塩基の配列と考えることもできる。その場合、塩基のU(ウラシル)をT(チミン)に置き換えて読む。
遺伝コードにおける塩基とアミノ酸の対応
タンパク質を構成する主要なアミノ酸は20種類ある。一方、DNAの構成要素であるヌクレオチドの塩基は、上記のようにわずか4種類である。アミノ酸20種類を区別して指定するのに、塩基1つでは4種類しか区別できず、また、塩基2つの組み合わせでも4×4 = 16種類しか区別できないので足りない。実際の生体内では3個ずつの塩基が1セットになって、アミノ酸1個に対応する形でタンパク質をコードしている。塩基3個の場合、理論的には、4×4×4 = 64種類を区別してコードすることが可能である。実際には、20種類のアミノ酸に加え、どのアミノ酸にも対応しないコドンもあり、ペプチド鎖合成の終了を意味している。これは終止コドンと呼ばれる。また、1つのアミノ酸は複数のコドンと対応している場合が多い。
生物種による利用コドンの偏り
RNAコドン表は、mRNA上にあるコドンとそれが指定するアミノ酸との関係を示した表である。
原核生物と真核生物など、生物の種類によって用いているコドンは下記のコドン表とは一部異なっている場合もある。
また、複数のコドンが対応しているアミノ酸では、生物種によって、また同種生物内でも遺伝子によって同義コドンを用いる頻度の傾向が大きく異なり、自己組織化写像などを用いることによってDNA断片から生物種を推定することが出来る。この頻度の違いをコドン出現頻度 (codon usage, codon frequency)の違いという。コドン出現頻度の違いは遺伝子の発現量やそのコドンに対応する tRNA の量と関係があることが知られている。発現量の多い遺伝子のコドン出現頻度の偏りは大きくなり、頻出するコドンに対応する tRNA は細胞内の存在量も多い。これは組換えタンパク質を本来の生物種とは異なる生物種で発現させる際などに問題になる。例えば、ある導入遺伝子に使われているコドンが、ホスト細胞では頻度の低いコドンである場合には、導入遺伝子産物の生産が少ないといったことが起こりうる。このような場合には導入遺伝子にサイレント突然変異を起こしコドンを最適化したり、導入細胞側にマイナー tRNA を過剰に発現させたりすると改善される場合もある。
遺伝コードの解読

The genetic code
DNAの構造がジェームズ・ワトソン、フランシス・クリック、モーリス・ウィルキンス、ロザリンド・フランクリンらによって解明されたあと、タンパク質が生体内でどのようにコードされているかということの解明に向けて真剣な努力が払われた。ジョージ・ガモフは、生体の細胞内でタンパク質をコードするのに用いられている20ほどの異なるアミノ酸を指定するのに3文字の暗号が用いられていると仮定した(なぜなら4nが少なくとも20以上であるようなnは3が最小だから)。コドンがまさにDNAの3塩基に対応しているという事実を最初に示したのはクリックとシドニー・ブレナーらの実験である [1]。はじめて一つのコドンを明らかにしたのは1961年、アメリカ国立衛生研究所のマーシャル・ニーレンバーグとハインリッヒ・マッタイであった [2]。彼らは無細胞系でポリウラシルRNA配列(これは生化学的記号でUUUUU....と表される)を翻訳した。合成できたポリペプチドはフェニルアラニンのみからなるものであることを発見した。このことから、コドンUUUがアミノ酸フェニルアラニンを指定すると推定した。ニーレンバーグと共同研究者らはこの研究を推し進めていって、個々のコドンのヌクレオチド組成を決定することができた。配列の順序を決定するのに3ヌクレオチドがリボソームに固定され、アミノアシルtRNAを放射線標識して、どのアミノ酸がコドンに対応するかを決定した。ニーレンバーググループは64コドン中54の配列を決定できた。続いてハー・ゴビンド・コラナが残りのコドンを決定することができた。その後程なくロバート・W・ホリー が翻訳の際のアダプター分子であるtRNAの構造を明らかにした。この研究は、1959年にRNA合成の酵素学に関する研究によってノーベル賞を受賞したセベロ・オチョアの初期の研究に基づいていた。1968年にコラナ、ホリー、ニーレンバーグらも生理学あるいは医学ノーベル賞を受賞した。
遺伝コードを介して情報を伝達する
生物のゲノムはDNA中に刻まれている。ウイルスの中にはゲノムがRNAに刻まれているものもある。ゲノム中で1つのタンパク質あるいは1つのRNAをコードしている部分を遺伝子という。タンパク質をコードしている遺伝子はコドンと呼ばれる3ヌクレオチドの単位から構成されており、各コドンは1つのアミノ酸をコードしている。コドンのサブユニットである各ヌクレオチドはさらにリン酸、デオキシリボース、窒素を含んだ4種類のヌクレオチド塩基のうちの1つ、という要素からなる。プリン塩基のアデニン(A)とグアニン(G)は大きな塩基で芳香環を2つもつ。ピリミジン塩基のシトシン(C)とチミン(T)は小さい塩基で芳香環を1つしかもたない。DNA鎖は2重らせん構造を取るとき、塩基対結合として知られる配置によって水素結合で互いに会合している。これらの結合はほとんど常に、一方の鎖のアデニンと他方の鎖のチミンの間、同じくシトシンとグアニンの間で行われる。これは2重らせん中のAとTの数、同様にGとCの数が同じであることを意味している。RNAの場合はチミン(T)の代わりにウラシル(U)が用いられ、デオキシリボースの代わりにリボースが用いられる。
タンパク質をコードする遺伝子はDNAに類縁のポリマーRNAである鋳型分子、メッセンジャーRNAあるいはmRNAに転写される。この分子は続いてリボソーム上でアミノ酸鎖つまりポリペプチドに翻訳される。翻訳プロセスは個々のアミノ酸に特異的なトランスファーRNAを必要とする。アミノ酸はtRNAに共有結合している。グアノシン3リン酸(GTP)がエネルギー源となり、一群の翻訳因子も必要である。tRNAはmRNAのコドンに相補的なアンチコドンをもっており、3'末端のCCAで共有結合によってアミノ酸を結合・保持する。各tRNAは特異的なアミノ酸をアミノアシルtRNA合成酵素によって結合・保持する。この酵素はアミノ酸と、対応するtRNAの双方に高い特異性をもっている。これらの酵素に高い特異性があることが、タンパク質の翻訳が厳密に行われることの主要な理由である。
3ヌクレオチドからなるトリプレットコドンによって可能なコドンの組合せは、43=64種類ある。実際、標準遺伝コードの64コドン全てがアミノ酸あるいは翻訳ストップシグナルに割り当てられている。例えばRNAの塩基配列がUUUAAACCCであったとしよう。読み枠は先頭のU(慣例により5'から3'とする)から始めてコドンを当てはめると3コドンが得られる。つまり、UUU、AAA、CCCである。各コドンは1つのアミノ酸に対応し、このRNAの塩基配列は3アミノ酸からなる配列に翻訳される。コンピュータ科学に比較対照されるものを求めると、コドンはワードに相当し、データ操作の標準的な単位であり(タンパク質のアミノ酸1つのように)、ヌクレオチド1つは1ビットに相当する。
標準遺伝コードが次の表に示されている。表1は64コドン各々がどのアミノ酸に対応するかを示す。表2は翻訳される標準的なアミノ酸20個の各々がどのコドンに対応するかを示す。これらは、それぞれ、コドン対照表およびコドン逆対照表と呼ばれる。例えばコドンAAUはアスパラギンに対応し、UGUとUGCはシステインに対応する(アミノ酸を標準的な3文字記号で表すとそれぞれAsnとCysである)。
RNAコドン表
| 2つ目の塩基 | |||||
|---|---|---|---|---|---|
| U | C | A | G | ||
| 1つ目の塩基 | U | UUU→Phe、 | UCU→Ser、 | UAU→Tyr、 | UGU→Cys、 |
| C | CUU→Leu、 | CCU→Pro、 | CAU→His、 | CGU→Arg、 | |
| A | AUU→Ile、 | ACU→Thr、 | AAU→Asn、 | AGU→Ser、 | |
| G | GUU→Val、 | GCU→Ala、 | GAU→Asp、 | GGU→Gly、 | |
| 3文字記号 | 1文字記号 | 呼称 | コドン |
|---|---|---|---|
| Ala | A | アラニン | GCU、GCC、GCA、GCG |
| Arg | R | アルギニン | CGU、CGC、CGA、CGG、AGA、AGG |
| Asn | N | アスパラギン | AAU、AAC |
| Asp | D | アスパラギン酸 | GAU、GAC |
| Cys | C | システイン | UGU、UGC |
| Gln | Q | グルタミン | CAA、CAG |
| Glu | E | グルタミン酸 | GAA、GAG |
| Gly | G | グリシン | GGU、GGC、GGA、GGG |
| His | H | ヒスチジン | CAU、CAC |
| Ile | I | イソロイシン | AUU、AUC、AUA |
| Leu | L | ロイシン | UUA、UUG、CUU、CUC、CUA、CUG |
| Lys | K | リシン | AAA、AAG |
| Met | M | メチオニン | AUG |
| Phe | F | フェニルアラニン | UUU、UUC |
| Pro | P | プロリン | CCU、CCC、CCA、CCG |
| Ser | S | セリン | UCU、UCC、UCA、UCG、AGU、AGC |
| Thr | T | トレオニン | ACU、ACC、ACA、ACG |
| Trp | W | トリプトファン | UGG |
| Tyr | Y | チロシン | UAU、UAC |
| Val | V | バリン | GUU、GUC、GUA、GUG |
| — | 開始コドン | AUG、(AUA)、(GUG) | |
| — | 終止コドン | UAG、UGA、UAA | |
重要な特徴
塩基配列の読み枠
コドンの割り当ては翻訳が開始される先頭のヌクレオチドから行われる。例えば塩基鎖がGGGAAACCCで先頭から読まれるとすると、コドンはGGG、AAA、CCCとなり、2番目から読まれるとすると、コドンはGGA、AAC、3番目から読まれるとすると、GAA、ACCとなる。この例ではコドンが部分的な場合は無視した。このように塩基配列がどうであれ、読み枠は3つ(の塩基)であり、各々異なるアミノ酸配列を生じる(この例では順に、Gly-Lys-Pro、Gly-Asp、Glu-Thrである)。2本鎖DNAには可能な読み枠は6つあり、一方の鎖に3つ読み枠があり(他方の鎖に)反対方向に3つある。
タンパク質のアミノ酸配列に翻訳される実際の読み枠は開始コドンによって割り当てられ、通常、それはmRNAの配列の最初のAUGコドンである。ヌクレオチド塩基が3の倍数以外の数だけ挿入されたり欠失を起こした場合に生ずる、読み枠が乱されるような突然変異はフレームシフト変異として知られる。このような突然変異は、たとえタンパク質として産生されても、その機能を損うため、生体内のタンパク質をコードしている配列の中でまれなものとなる。しばしばそのような誤って作られたタンパク質はタンパク質分解性の崩壊プロセスのターゲットとなる。加えてフレームシフト突然変異は往々にして終止コドンを生じ、タンパク質産生を中途終止させる(例[2])。次代に遺伝するフレームシフト突然変異がまれな理由は、もし翻訳されるタンパク質が、その生物が直面する選択圧のもとで生育に必須なものであるとしたら、機能をもったタンパク質が存在しないことによって、その生物が生存する以前に致死となるかも知れないからである。
開始コドン、終止コドン
翻訳は核酸鎖の開始コドンから始まる。終止コドンと違って、開始コドンだけでは翻訳プロセスが始められるには十分でない。開始コドン近くの配列の条件や開始因子も翻訳開始に必要である。最も一般的な開始コドンはAUGであり、これはメチオニンをコードするため、アミノ酸鎖の先頭で最も多いのはメチオニンである。終止コドンは3つあってそれぞれ名称がある:UAGはアンバー(amber)、UGAはオパール(opal)(ときにアンバーumberと呼ばれる)、UAAはオーカー(ochre)。「アンバーamber」は発見者Richard EpsteinとCharles Steinbergによって彼らの友人Harris Bernsteinがファミリー名をドイツ語でamberということに因んで命名された。他の2つの終止コドンは色彩名をつける原則によって命名された。終止コドンは停止コドンとも呼ばれこれら終止シグナルコドンに相補的なアンチコドンをもった対応するtRNAというのはないが、解離因子を結合させることによって、作られたばかりのポリペプチドをリボソームから解離するシグナルとなる。
遺伝コードの縮重
遺伝コードは冗長であるが多義性はない(上掲のコドン表で全ての対応を見よ)。例えばコドンGAAとGAGはどちらもグルタミン酸を指定するが(冗長性)、どちらも他のアミノ酸を指定するということはない(非多義性)。一つのアミノ酸をコードするコドンは3つのヌクレオチドのうちどこかで異なる場合がある。例えば、グルタミン酸はコドンGAA、GAGによって指定されるが(第3番目の位置でヌクレオチドが異なる)、ロイシンはコドンUUA、UUG、CUU、CUC、CUA、CUGによって指定されるし(先頭と3番目の位置で異なる)、セリンはコドンUCA、UCG、UCC、UCU、AGU、AGCによって指定される(先頭、2番目、3番目の位置で異なる)。コドンのヌクレオチドの3つの位置の一つで異なるヌクレオチドによって同じアミノ酸が指定される場合、4重に縮重していると言われる。例えばグリシンのコドン(GGA、GGG、GGC、GGU)の塩基の第3番目の位置はこの位置でのヌクレオチドの置換全てが同義であるため、つまり、対応するアミノ酸に変化を起こさないため4重に縮重した位置である。コドンのうち3番目の位置のみで4重に縮重したものがある。コドンの3つの位置のうち一つであり得る4種のヌクレオチドの2つのみで同じアミノ酸が指定される場合2重に縮重していると言われる。例えばグルタミン酸コドンGAA、GAGの3番目の位置は2重に縮重しており、ロイシンコドン(UCA、UCC、CCU、CCC、CCA、CCG)の先頭位置も同じである。2重に縮重した位置においては同義性ヌクレオチドは常に何れもがプリンであるか(A/G)、ピリミジンであるか(C/U)であるため、2重に縮重した位置ではトランスバージョン置換(プリンからピリミジンあるいはその逆)のみが非同義である。コドンの3つの位置のいずれかでヌクレオチド置換によってアミノ酸が変化する場合、その位置は縮重がないといわれる。3重に縮重した位置は1つだけあって、4つのヌクレオチドのうち3つの変化がアミノ酸に変化をもたらさないが、残りの1つのヌクレオチドに変わるとアミノ酸が変わる。これはイソロイシンコードの3番目の位置である。AUU、AUC、AUAは全てイソロイシンをコードするが、AUGはメチオニンをコードする。計算上はこの位置はしばしば2重縮重位置として扱う。[4]
6つの異なったコドンでコードされているアミノ酸は3つある:セリン、ロイシン、アルギニンである。ただ1つのコドンで指定されているアミノ酸は2つだけある。1つはメチオニンで、コドンAUGで指定され、これは翻訳の開始も指定する。もう1つはトリプトファンでコドンUGGで指定される。遺伝コードの縮重はサイレント突然変異の存在を裏付ける。
縮重があるのはトリプレットコードが20のアミノ酸と1つの終止コドンを指定するからである。塩基が4つあるトリプレットコドンで少なくとも21の異なったコードを実現しなければならない。例えばコドンが2つの塩基だったら16アミノ酸しかコードできない(42=16であるから)。少なくとも21コード必要なので43=64のコドンが実現できてしまうことになって、縮重が起こるのが当然となる。
遺伝コードはこのような性質によって点突然変異のようなエラーに堪えるものとなっている。例えば、理論上4重縮重のあるコドンは3番目の位置の点突然変異がどのように起こっても問題はない。実際は多くの生物でコドンの利用の偏りがこのことに制限を与えるが。2重縮重のあるコドンは3番目の位置の可能な3つの点突然変異のうち1つが起こっても問題はない。トランジション突然変異(プリンからプリンへの、あるいはピリミジンからピリミジンへの突然変異)のほうがトランスバージョン突然変異(プリンからピリミジンへの突然変異、あるいはその逆)よりも起こりやすいから、このような2重縮重位置でのプリンの同等性あるいはピリミジンの同等性は、エラーに強い性質が付け加わることになる。
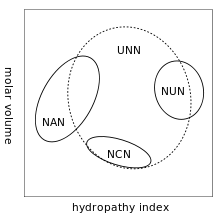
アミノ酸残基の分子量(縦軸)と疎水親水性(横軸)でグループ分けしたコドン
冗長性のもたらす実際上の結果は、エラーが遺伝コードに起こってもそれはサイレントであって、同じアミノ酸への置換しか起こさないから、タンパク質が変化して疎水性や親水性に変化を及ぼすというようなことはなく、タンパク質に影響の及ばないエラーであるということである。例えばNUN(Nはヌクレオチドを示す)というコドンは親水性のアミノ酸をコードする傾向がある。NCNはアミノ酸残基の大きさが小さく疏水親水性が中間的であり、NANは平均サイズの親水性アミノ酸残基、UNNは非親水性のアミノ酸残基をコードする。[5][6]
そうは言っても点突然変異が起こると機能の損われたタンパク質が作られる可能性がある。ヘモグロビン遺伝子に突然変異が起こって鎌状赤血球症が起こされる例を取り挙げてみよう。この点突然変異では親水性のグルタミン酸(Glu)が1ヵ所疎水性のバリン(Val)に置き換わっており、β-グロビンの可溶性が低下している。この場合には、突然変異によって、ヘモグロビンは、バリンのグループ間の疎水性相互作用が変化し、それが原因となって直鎖ポリマーとなり、赤血球は鎌状細胞に変形する。鎌状赤血球症は一般に新規の突然変異によっては起こらない。むしろ、マラリア常在地域においてこの遺伝子ヘテロの人々がマラリアのPlasmodium寄生体にいくほどかの抵抗性(ヘテロ体の有利さ)をもつことによって、自然選択作用によって存続している(サラセミアと同様なやり方である)。
このようにアミノ酸に対するコードに変化がもたらされる理由は、tRNAのアンチコドン1番目の塩基が修飾されることにある。こうして形成される塩基対はゆらぎ塩基対と呼ばれる。修飾される塩基はイノシンであったり非Watson-Crick対であるU-G塩基対であったりする。
標準遺伝コードの変形
標準遺伝コードにはわずかな変動があるだろうということは早くから予見されていたが、[7]1979年までは発見されなかった。同年、ヒトミトコンドリア遺伝子の研究者が異なるコードを発見した。以来、わずかに変形したものが数多く発見された。[8] それらは種々のミトコンドリアのコードであったり、[9]Mycoplasmaの、コドンUGAをトリプトファンに翻訳するようなわずかな変更の見られるものであった。細菌と古細菌ではGUGとUUGが共通する開始コドンである。珍しい例では、同じ種でも特定のタンパク質で、通常使われるのと異なる開始コドンが使われる場合がある。[8]
タンパク質の中には、mRNA上のシグナル配列に変動があり、それに伴って標準的な終止コドンに他の非標準的なアミノ酸が置き換っている場合がある。関連文献で議論されているように、UGAはセレンシステインをコードし、UAGはピロリジンをコードしている場合がある。セレンシステインは現在、21番目のアミノ酸と見なされており、ピロリジンは22番目のアミノ酸と見なされている。遺伝コードの変形の詳細はNCBIウェブサイトで見ることができる。[8]
これまでに知られたコードにはこのような違いはあるにせよ、それらの間には顕著な共通性が見られるし、総ての生物でこのコード機構は同じであると考えられる。つまり、3塩基コドンであり、tRNA、リボソームを必要とし、コード読み取り方向は同じであり、コードの3文字を一度に翻訳してアミノ酸に変える点である。
遺伝コードの起源についての理論
地球上の生命体によって用いられている遺伝コードには変形は見られるにせよ互いによく似ている。地球上の生命体にとって、同様な利用価値のある遺伝コードはほかに多くの可能性があるのだから、進化論的には、生命の歴史のきわめて初期に遺伝コードが確立したことが、次のことを考慮しても示唆される。tRNAの系統学的解析によって、今日のアミノアシルtRNA合成酵素のセットが存在する以前にtRNA分子が進化してきたと推定された。[10]
遺伝コードはアミノ酸へのランダムな対応ではない。[11]例えば同じ生合成経路に関与するアミノ酸はコドンの第1塩基が同じ傾向がある。[12]物理的性質の似たアミノ酸はよく似たコドンに対応している傾向がある。[13][14]
遺伝コードの進化を説明しようとしている多くの理論に貫かれている3つのテーマがある(3つのパターンはこれが起源である)。[15]1つは最近のアプタマー(リガンド結合能のあるオリゴヌクレオチド)実験で説明されている。アミノ鎖の中にはコードする3塩基トリプレットに選択的な化学的親和性をもっているものがある。[16]これは、現在のtRNAと関連酵素によって行われている複雑な翻訳機構は後代になって発達してきたものであって、元々はタンパク質のアミノ酸配列は塩基配列を直接の鋳型としていたことを示唆する。もう一つは、今日われわれが目にする標準遺伝コードはもっと簡単なコードから生合成的な拡張プロセスを経て発達したと考える。この考えは、原始生命体は新しいアミノ酸を(例えば代謝の副産物として)発見し、のちに遺伝コードの機構に組み入れて行った、とする。現在に比べ過去にはアミノ酸は種類が少なかったと示唆される状況証拠は沢山あるが、[17]どのアミノ酸がどういう順でコードに入れられたかの正確かつ詳細な仮説は議論が大きく分かれている。[18][19]なお、2018年1月現在、チロシンとトリプトファンについては、20-24億年前の酸素増大イベント(大酸化イベント)に耐えるために獲得された可能性を、量子化学計算と生化学実験から提示した研究が発表されており、アミノ酸の機能的特性が遺伝暗号を決定づけていたことを示唆している。[20]3番目は、遺伝コードでのコードの割り当ては、突然変異の効果が最小となるように自然選択が作用してなされたとする。[21]
引用文献
^ Crick FH, Barnett L, Brenner S, Watts-Tobin RJ. 1961. General nature of the genetic code for proteins. Nature 192:1227-32.
^ Nirenberg MW, Matthaei JH. 1961. The dependence of cell-free protein synthesis in E. coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA 47:1588-602.
^ コドンAUGにはメチオニンに対するコードとしての働きと翻訳開始位置としての働きがある。mRNAのコード領域において初めてAUGが現れるとタンパク質への翻訳が開始される。
^ How nonsense mutations got their names
^ Yang et al. 1990. In Reaction Centers of Photosynthetic Bacteria. M.-E. Michel-Beyerle. (Ed.) (Springer-Verlag, Germany) 209-218
^ Genetic Algorithms and Recursive Ensemble Mutagenesis in Protein Engineering http://www.complexity.org.au/ci/vol01/fullen01/html/
^ Crick, F. H. C. and Orgel, L. E. (1973) "Directed panspermia." Icarus 19:341-346. p. 344: "It is a little surprising that organisms with somewhat different codes do not coexist." (Further discussion at [1])- ^ abcNCBI: "The Genetic Codes", Compiled by Andrzej (Anjay) Elzanowski and Jim Ostell
^ Jukes TH, Osawa S, The genetic code in mitochondria and chloroplasts., Experientia. 1990 Dec 1;46(11-12):1117-26.
^ De Pouplana, L.R.; Turner, R.J.; Steer, B.A.; Schimmel, P. (1998). “Genetic code origins: tRNAs older than their synthetases?”. Proceedings of the National Academy of Sciences 95 (19): 11295. doi:10.1073/pnas.95.19.11295. PMID 9736730. http://www.pnas.org/cgi/content/full/95/19/11295.
^ Freeland SJ, Hurst LD (September 1998). “The genetic code is one in a million”. J. Mol. Evol. 47 (3): 238--48. doi:10.1007/PL00006381. PMID 9732450. http://link.springer-ny.com/link/service/journals/00239/bibs/47n3p238.html.
^ Taylor FJ, Coates D (1989). “The code within the codons”. BioSystems 22 (3): 177--87. doi:10.1016/0303-2647(89)90059-2. PMID 2650752.
^ Di Giulio M (October 1989). “The extension reached by the minimization of the polarity distances during the evolution of the genetic code”. J. Mol. Evol. 29 (4): 288--93. doi:10.1007/BF02103616. PMID 2514270.
^ Wong JT (February 1980). “Role of minimization of chemical distances between amino acids in the evolution of the genetic code”. Proc. Natl. Acad. Sci. U.S.A. 77 (2): 1083--6. doi:10.1073/pnas.77.2.1083. PMID 6928661.
^ Knight, R.D.; Freeland S. J. and Landweber, L.F. (1999) The 3 Faces of the Genetic Code.Trends in the Biochemical Sciences 24(6), 241-247.
^ Knight, R.D. and Landweber, L.F. (1998). Rhyme or reason: RNA-arginine interactions and the genetic code.Chemistry & Biology 5(9), R215-R220. PDF version of manuscript
^ Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code. Molecular Biology and Evolution 19, 1645-1655.
^ Amirnovin R. (1997) An analysis of the metabolic theory of the origin of the genetic code.Journal of Molecular Evolution 44(5), 473-6.
^ Ronneberg T.A.; Landweber L.F. and Freeland S.J. (2000) Testing a biosynthetic theory of the genetic code: Fact or artifact?Proceedings of the National Academy of Sciences, USA 97(25), 13690-13695.
^ Moosmann, Bernd; Irimie, Florin-Dan; Toşa, Monica Ioana; Hajieva, Parvana; Granold, Matthias (2018-01-02). “Modern diversification of the amino acid repertoire driven by oxygen” (英語). Proceedings of the National Academy of Sciences 115 (1): 41–46. doi:10.1073/pnas.1717100115. ISSN 1091-6490. PMC PMC5776824. PMID 29259120. https://www.pnas.org/content/115/1/41.
^ Freeland S.J.; Wu T. and Keulmann N. (2003) The Case for an Error Minimizing Genetic Code.Orig Life Evol Biosph. 33(4-5), 457-77.
参考文献
- 蛋白質核酸酵素 51(7), 2006:遺伝暗号解読40周年 西村暹、三浦謹一郎編 共立出版 ISSN 0039-9450
関連項目
- アンチコドン
- tRNA
- mRNA
- rRNA
- タンパク質を構成するアミノ酸
- 開始コドン
- 終止コドン
外部リンク
コドンと翻訳・蛋白合成 - ビジュアル生理学 内の項目
遺伝暗号(コドン)使用の種による多様性 - 遺伝子電子館、国立遺伝学研究所
| |||||||||||||||||||||||
